以下文章来源于实验老司机 ,作者煲仔饭
单碱基编辑(Base Editing)是一种能够在基因组中直接实现碱基替换的技术,无需依赖DNA双链断裂(DSB)和同源重组修复。与CRISPR-Cas9系统相比,该技术具有更高的精确性和更低的脱靶风险,广泛应用于遗传疾病模型构建、功能基因组学研究及潜在基因治疗领域。
1 技术原理
单碱基编辑系统的核心由三个功能模块组成:
1. Cas9变体蛋白:通常使用dCas9(完全失去核酸酶活性)或nCas9(保留单链切口酶活性),通过向导RNA(sgRNA)靶向定位至目标DNA区域。
2. 脱氨酶:
胞嘧啶脱氨酶(如rAPOBEC1):在CBE(Cytosine Base Editor)系统中催化胞嘧啶(C)转化为尿嘧啶(U),后续DNA复制中U被识别为胸腺嘧啶(T),实现C→T(或G→A)的转换。
腺嘌呤脱氨酶(如TadA*变体):在ABE(Adenine Base Editor)系统中催化腺嘌呤(A)转化为肌苷(I),复制后I被识别为鸟嘌呤(G),实现A→G(或T→C)的转换。
3. DNA修复抑制元件:尿嘧啶糖苷酶抑制剂(UGI)可阻断尿嘧啶的切除修复,提高编辑效率。
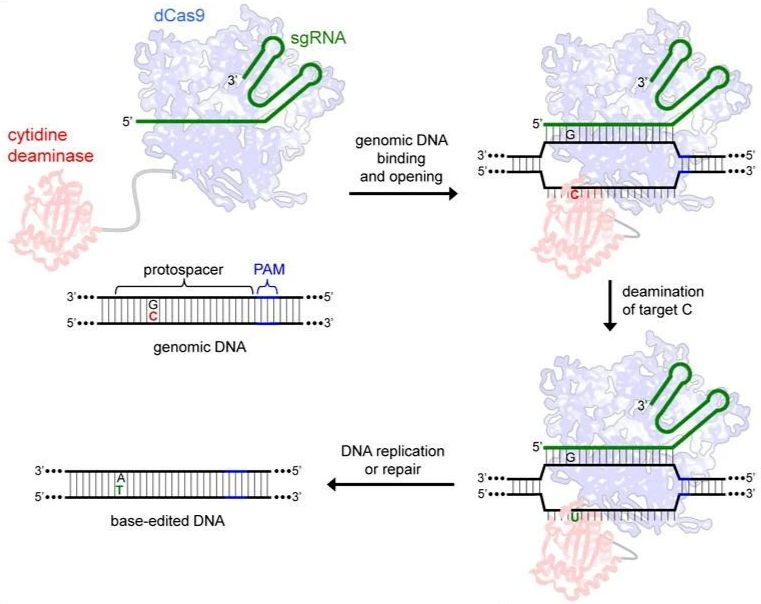
碱基编辑策略:在由向导 RNA(绿色)指定的位点处具有靶标 C(红色)的 DNA 由 dCas9(蓝色)结合,从而介导局部 DNA 链分离。通过束缚的 APOBEC1 酶(红色)进行的胞苷脱氨将单链靶标 C→U 转化。
图片来源:doi: 10.1038/nature17946。
编辑过程:
1. sgRNA引导Cas9蛋白结合至目标DNA区域,形成R-loop结构,暴露单链DNA。
2. 脱氨酶对单链DNA中的特定碱基(C或A)进行化学修饰。
3. DNA复制或修复过程中,修饰后的碱基被固定为永久性改变。
2 实验材料

3 实验步骤
3.1 靶点设计与sgRNA合成
3.1.1 靶点筛选与验证
1. 编辑窗口的定位规则:
(1)坐标定义:
以sgRNA的5'端第一个核苷酸为第1位,向3'端依次编号
PAM序列(NGG)位于sgRNA的3'端下游,不计入sgRNA的20 bp序列
(2)不同编辑器的活性窗口:

(3)实验验证方法:
合成包含目标位点的DNA片段(如gBlock),通过体外编辑实验测定窗口效率
使用DeepBaseEdit预测活性窗口
2. 设计工具:
使用CRISPOR或Benchling输入目标基因序列(如NCBI Gene ID),设置以下参数:
PAM类型:NGG(适用于SpCas9变体)
sgRNA长度:20 bp(不含PAM)
编辑窗口优先级:
CBE系统:sgRNA第4-8位(以PAM远端为第1位)
ABE系统:sgRNA第4-7位
脱靶分析:勾选“全基因组脱靶扫描”,排除与其它基因同源性>80%的候选sgRNA
3. 示例靶点:
假设目标位点为人类HBB基因c.20A>T突变(镰刀型细胞贫血):
靶序列:5'-GACGTGCGACTGAGGAGAAG[T]CTGC-3'(方括号内为突变位点)
设计sgRNA:5'-GACTGAGGAGAAGTCTGCGG-3'(下划线为编辑窗口,PAM为GG)
3.1.2 sgRNA oligo合成与退火
1. 合成要求:
上游引物(含Overhang):5'-CACCG**N20**-3'(N20为sgRNA靶序列,不含PAM)
下游引物:5'-AAAAC**N20rev**-3'(N20rev为sgRNA靶序列反向互补)
2. 退火反应:
配制退火混合液(20 μL体系):

3. 程序设置:95℃ 5min → 每分钟降温1℃至25℃ → 4℃保存
4. 稀释退火产物:用无菌水1:10稀释备用
3.1.3 sgRNA载体连接与验证
1. 载体酶切:使用BsmBI限制性内切酶(Thermo Fisher, #ER0451)切割pU6-sgRNA载体:
反应体系(50 μL):

37℃孵育2h → 琼脂糖凝胶电泳回收线性化载体
2. 连接反应:
体系(10 μL):

bl3感受态细胞
3. 阳性克隆筛选:
挑取单菌落接种于LB培养基(含Amp抗性),37℃摇菌12h
提取质粒(使用Miniprep Kit)
Sanger测序验证(引物:U6-F: 5'-GAGGGCCTATTTCCCATGATT-3')
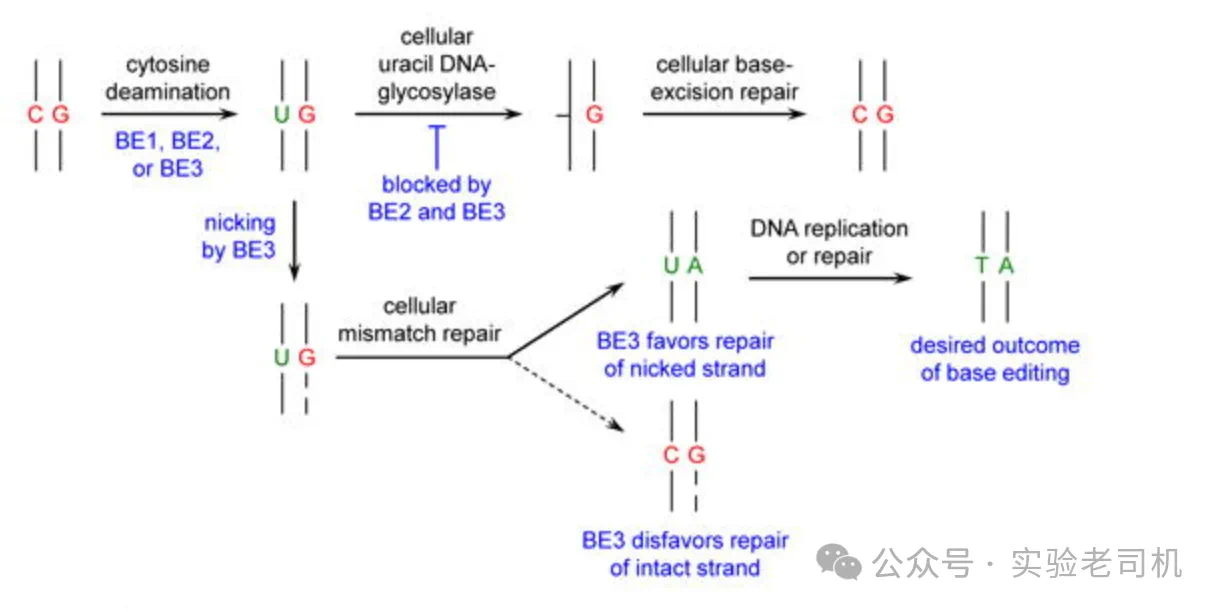
哺乳动物细胞中可能的碱基编辑结果:初始编辑导致 U:G 错配。尿嘧啶 DNA 糖基化酶 (UDG) 对 U 的识别和切除启动碱基切除修复 (BER),从而导致恢复到 C:G 起始状态。BE2 和 BE3 会阻碍 BER,从而抑制 UDG。U:G 错配也由错配修复 (MMR) 处理,错配修复优先修复错配的缺口链。BE3 缺口包含 G 的未编辑链,有利于将 U:G 错配解决为所需的 U:A 或 T:A 结果。
图片来源:doi: 10.1038/nature17946。
3.2 细胞转染
3.2.1 细胞培养前处理
1. 培养基配置(以HEK293T为例):
DMEM高糖培养基 + 10% FBS + 1%青霉素/链霉素
细胞传代:当汇合度达80-90%时,用0.25%胰蛋白酶-EDTA消化3min
2. 铺板参数:
6孔板:2×10^5 cells/孔(转染前24h铺板)
培养条件:37℃, 5% CO2, 湿度>90%
3.2.2 转染复合物制备(脂质体法)
1. 试剂配制(以Lipofectamine 3000为例):
Tube A(DNA混合液):

Tube B(脂质体混合液):
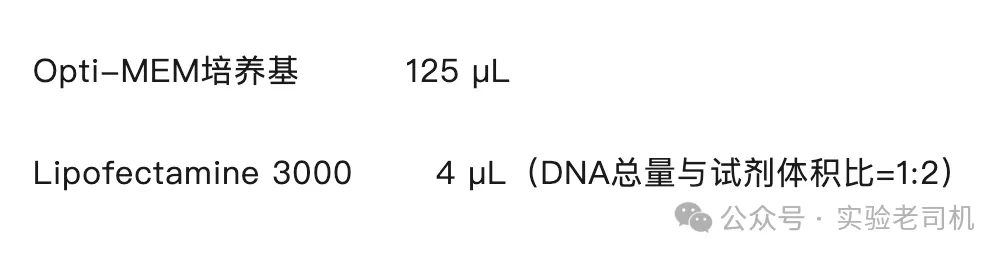
2. 孵育条件:
将Tube A和Tube B分别涡旋5s
将Tube B缓慢加入Tube A,轻柔吹打混匀
室温静置15min(形成DNA-脂质体复合物)
3.2.3 转染
1. 吸除细胞培养基,加入1.5 mL新鲜培养基(不含抗生素)
2. 将250 μL转染复合物逐滴加入孔板,十字摇匀
3. 6h后更换为完全培养基(含抗生素)
4. 转染24h后:加入嘌呤霉素(终浓度1-2 μg/mL)
5. 每日观察细胞状态:
正常:细胞贴壁良好,增殖减缓但无大面积死亡
异常:若24h内细胞死亡率>90%,需降低筛选浓度
6. 筛选72h后:更换为正常培养基
3.2.4 终止培养与细胞收集
1. 转染后72小时(完成嘌呤霉素筛选),弃去培养基
2. 加入1×PBS(预冷至4℃)轻柔洗涤细胞2次(每次2 mL/孔,6孔板)
3. 加入0.25%胰蛋白酶-EDTA(含酚红)1 mL/孔,37℃孵育2-3分钟
4. 显微镜下确认细胞脱离后,加入2 mL完全培养基终止消化
5. 将细胞悬液转移至1.5 mL离心管,1000 rpm离心5分钟
6. 弃上清,细胞沉淀用200 μL PBS重悬备用
3.3 编辑效率检测
3.3.1 PCR扩增
1. 取适量细胞沉淀,使用试剂盒提取基因组DNA(如QuickExtract DNA Extraction Solution(Epicentre))
2. 引物设计:引物距离编辑位点至少50 bp(避免引物二聚体干扰)
示例引物:
Forward: 5'-CCTGTACCACAGCCTTAAGG-3'
Reverse: 5'-GTAGTCGGTGTCGGCTGATA-3'
3. 配制PCR反应体系(25 μL体系):
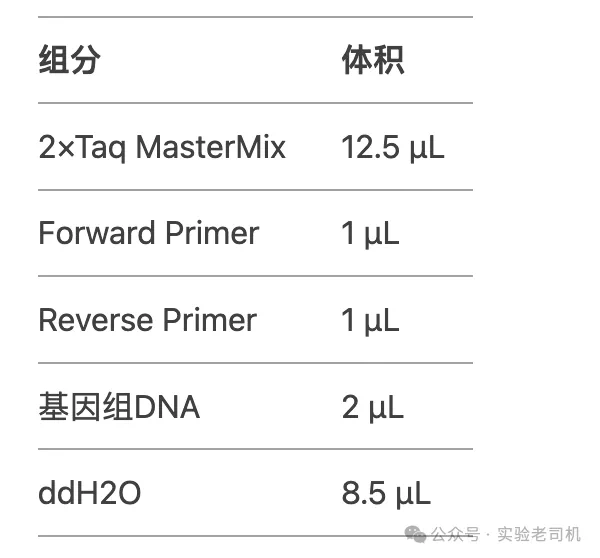
4. 扩增程序:
预变性:95℃ 3min
35个循环:95℃ 30s → 60℃ 30s → 72℃ 45s
终延伸:72℃ 5min
3.3.2 T7E1酶切分析
1. 异源双链制备:
混合PCR产物(200 ng)与等量野生型DNA
95℃变性5min → 每分钟降温2℃至25℃
2. 酶切反应:
反应体系:

37℃孵育30分钟 → 加入6×Loading Buffer终止反应
3. 电泳分析:2%琼脂糖凝胶,120 V电泳30分钟
切割条带比例估算公式:
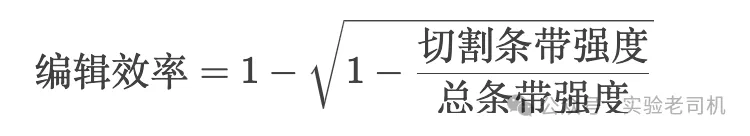
3.3.3 Sanger测序定量
1. PCR产物送测序,使用EditR(https://moriaritylab.shinyapps.io/editr_v10/)分析峰图
2. 计算公式:编辑效率=突变峰高度/(野生峰高度+突变峰高度)×100%
4 注意事项
4.1 sgRNA合成失败排查
无连接产物:检查BsmBI酶切效率(电泳确认载体线性化)
测序不匹配:重新设计引物避免回文结构
4.2 转染效率提升
低效处理:
增加DNA总量至3 μg/孔(需保持BE:sgRNA=3:1)
改用PEI转染(jetOPTIMUS®,Polyplus-transfection)
细胞毒性处理:
若转染后24小时细胞圆缩率>30%,降低脂质体用量20%
4.3 多靶点共转染
需控制总DNA量≤4 μg/孔
不同sgRNA质粒需使用不同抗性筛选标记(如sgRNA1用puromycin,sgRNA2用hygromycin)
5 参考资料
Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., & Liu, D. R. (2016). Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature, 533(7603), 420-424.
Gaudelli, N. M., Komor, A. C., Rees, H. A., Packer, M. S., Badran, A. H., Bryson, D. I., & Liu, D. R. (2017). Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature, 551(7681), 464-471.
Koblan, L. W., et al. (2021). Improving cytidine and adenine base editors by expression optimization and ancestral reconstruction. Nature Biotechnology, 39(5), 588-596.
Newby, G. A., & Liu, D. R. (2023). Base editing design guidelines using systematic target-sequence analysis. Nature Biotechnology, 41(5), 653-665.
Richardson, C. D., et al. (2016). Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology, 34(3), 339-344.
Brinkman, E. K., et al. (2014). Easy quantitative assessment of genome editing by sequence trace decomposition. Nucleic Acids Research, 42(22), e168.
Doench, J. G., et al. (2016). Optimized sgRNA design to maximize activity and minimize off-target effects of CRISPR-Cas9. Nature Biotechnology, 34(2), 184-191.
Ran, F. A., et al. (2013). Genome engineering using the CRISPR-Cas9 system. Nature Protocols, 8(11), 2281-2308.