RIP实验(RNA Binding Protein Immunoprecipitation Assay,RNA 结合蛋白免疫沉淀)是研究细胞内RNA与蛋白质结合的技术,是了解转录后调控网络动态过程的有力工具,可应用于研究miRNA调节靶点、RNA与RBPs(RNA结合蛋白)的互作关系等。
1. 实验原理
RIP可以看成是普遍使用的染色质免疫沉淀ChIP技术的类似应用,主要是运用针对目标蛋白的抗体或表位标记物捕获细胞核内或细胞质中内源性的RNA结合蛋白,通过免疫沉淀把RNA结合蛋白及其结合的RNA一起分离出来,将复合物中的RNA纯化出来后就可以对结合在复合物上的RNA 进行q-PCR验证或者测序分析。
2. 实验流程图
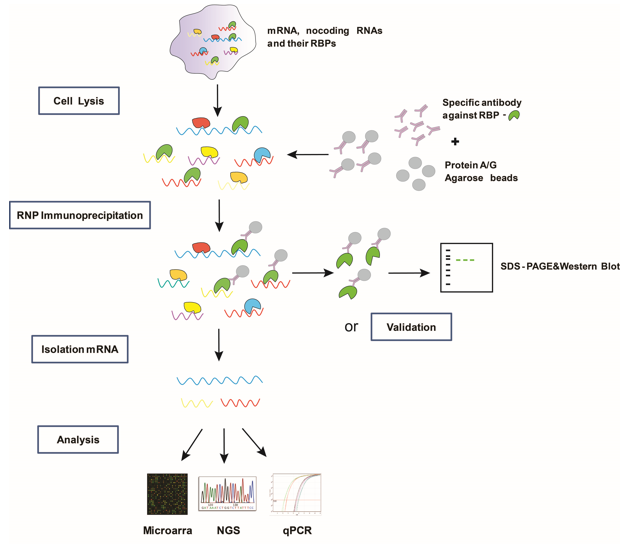
3. 实验步骤
3.1 获取细胞裂解液
3.1.1 冷PBS清洗培养皿或培养瓶中的细胞两次。
3.1.2 加入冷PBS后用细胞刮将细胞刮下来,收集至离心管中。
3.1.3 1500 rpm,4 ℃离心5 min,弃上清,收集细胞。
3.1.4 用与细胞等体积的RIP裂解液重悬细胞,吹打均匀后于冰上静置20 min。
3.1.5 每管分装200 μL细胞裂解液,贮存于-80 ℃。
3.2 制备磁珠-抗体
3.2.1 吸取50 μL 重悬后的磁珠悬液于每个离心管中,每管加入500 μL RIP Wash Buffer,涡旋震荡。
3.2.2 将离心管置于磁力架上,去上清,重复洗涤一次。
3.2.3 用100 μL的RIP Wash Buffer重悬磁珠,加入适量抗体,室温孵育30 min。
3.2.4 将离心管置于磁力架上,弃上清加入500 μL RIP Wash Buffer,涡旋震荡后弃上清,重复一次。
3.2.4 将离心管置于磁力架上,加入500 μL RIP Wash Buffer,混匀后置于冰上备用。
3.3 RNA结合蛋白免疫沉淀
3.3.1 将3.2.4的离心管放磁力架上,去上清,每管加入900 μL RIP Immunoprecipitation Buffer
3.3.2 迅速解冻3.1.5制备的细胞裂解液,离心,吸取100 μL上清液于磁珠-抗体复合物中,使得总体积为1 mL。
3.3.3 4 ℃孵育3 h或过夜孵育。
3.3.4 短暂离心,将离心管放在磁力架上,弃上清。
3.3.5 加入500 μL RIP Wash Buffer,涡旋震荡后将离心管放在磁力架上,弃上清,重复清洗3-5次。
3.4 RNA纯化
3.4.1 用150 μL Proteinase K Buffer重悬上述磁珠-抗体复合物,55 ℃孵育30 min。
3.4.2 孵育完之后,将离心管置于磁力架上,将上清液吸入一新的离心管中。
3.4.3 于每管上清液中加入250 μL RIP Wash Buffer,再加入400 μL苯酚:氯仿:异戊醇,涡旋震荡,室温下14 000 rpm离心10 min。
3.4.4 小心吸取350 μL上层水相,吸入另一新的离心管,于每管加入400 μL氯仿,涡旋震荡15 s,室温下14 000rpm离心10 min。
3.4.5 小心的吸取300 μL上层水相,吸入另一新的离心管,每管加入50 μL Salt SolutionⅠ,15 μL Salt SolutionⅡ,5 μL Precipitate Enhancer,850 μL无水乙醇(无RNase)混合,-80 ℃保持1 h至过夜,14 000 rpm,4 ℃离心30 min,小心去上清。
3.4.6 用80%乙醇冲洗一次,14 000 rpm,4 ℃离心15 min,小心去上清,空气中晾干,用10-20 μL DEPC水溶解,-80℃保存,可用于后续q-PCR验证或者测序分析。
4. 结果示例
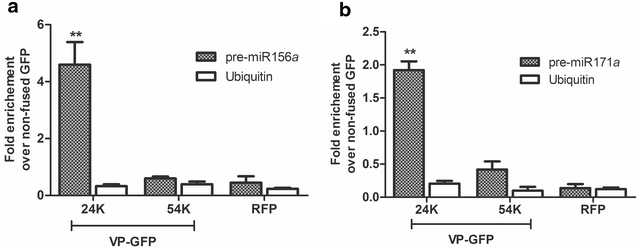
RIP 分析与本氏烟草中柑橘银屑病病毒 (CPsV) 的 24K 或 54K GFP 融合蛋白相关的前体。 进行 RT-qPCR 以确定本氏烟草 pre-miR156a (a) 或 pre-miR171a (b) 的积累水平
参考文献
[1]. Gagliardi M, Matarazzo MR. RIP: RNA Immunoprecipitation. Methods Mol Biol. 2016;1480:73-86.
[2]. Jayaseelan S, Doyle F, Tenenbaum SA. Profiling post-transcriptionally networked mRNA subsets using RIP-Chip and RIP-Seq. Methods. 2014 May 1;67(1):13-9.
[3]. Zambelli F, Pavesi G. RIP-Seq data analysis to determine RNA-protein associations. Methods Mol Biol. 2015;1269:293-303.
[4]. Nicholson CO, Friedersdorf MB, Bisogno LS, Keene JD. DO-RIP-seq to quantify RNA binding sites transcriptome-wide. Methods. 2017 Apr 15;118-119:16-23.