qPCR(Quantitative Real-time PCR)即实时荧光定量PCR技术,利用插入性染料或荧光探针,监控PCR过程中的荧光强度以比较样品间的DNA水平,最后通过标准曲线对未知模板进行定量分析的方法。根据化学发光原理可以分为:荧光染料法,TaqMan探针法。
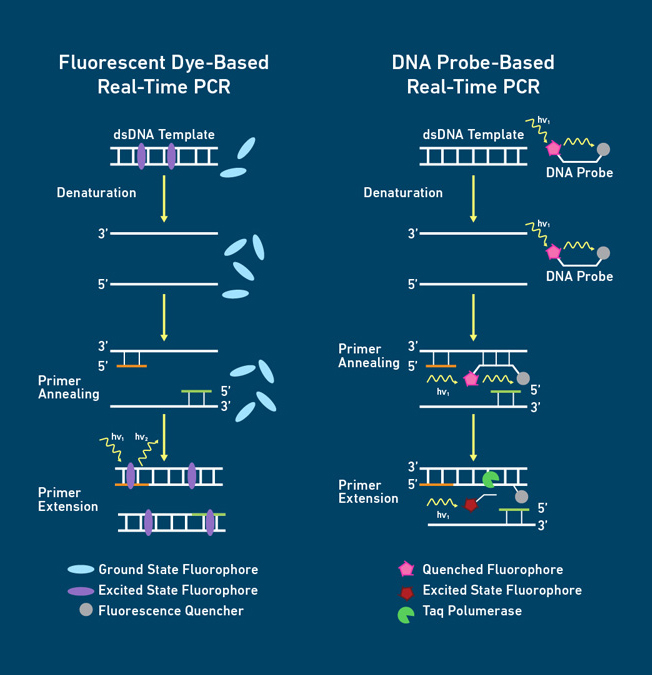
1. 实验原理
1.1 荧光染料法
在PCR反应体系中,加入过量荧光染料,荧光染料特异性地掺入DNA双链后,发射荧光信号,而不掺入链中的染料分子不会发射任何荧光信号,从而保证荧光信号的增加与PCR产物的增加完全同步。
1.2 TaqMan探针法
在PCR扩增时加入一对引物的同时另外加入一条特异性的TaqMan荧光探针,该探针与模板特异性地结合,其结合位点在两条引物之间。当探针完整时,报告荧光基团和淬灭荧光基团的空间距离很近,报告荧光基团发射的荧光信号会被淬灭荧光基团吸收,使仪器检测不到荧光信号。在PCR延伸阶段,Taq DNA 聚合酶沿着模板链从5'到3'的方向合成新链,当Taq DNA 聚合酶到达探针结合位点时,Taq DNA 聚合酶的5'-3'核酸外切酶活性将探针5'端连接的报告荧光基团切割下来,使报告荧光基团和淬灭荧光基团分离,从而发出荧光,切割的荧光分子数与PCR产物的数量成正比,因此,通过检测PCR反应体系中的荧光强度可以达到检测PCR产物扩增量的目的。
2. 实验步骤
2.1 总RNA抽提
2.1.1细胞裂解或组织匀浆
贴壁细胞:吸尽培养基,一般六孔板每孔加1 mL Trizol,12孔板每孔加0.5 mL Trizol。晃动3-5下,再用移液器吹打数次,确保全部裂解,然后转移至1.5 mL离心管中。
悬浮细胞:离心收集细胞,吸尽液体,每 5×106 细胞加入1 mL Trizol。用枪吹打确保全部裂解。
组织:组织块放入研钵中,加入少量液氮,迅速研磨,带组织变软,再加少量液氮,再研磨,如此三次,按每50-80 mg组织加入1 mL Trizol,转移入离心管。加入Trizol的裂解液室温放置5 min,4 ℃ 12 000 rpm离心10 min,吸取澄清的Trizol裂解产物(上清液)至新的离心管中,去除不溶物或油脂状漂浮物。
2.1.2 每使用1 mL Trizol加入0.2 mL氯仿,剧烈振荡15 s,室温放置3 min,4 ℃ 12 000 rpm离心15 min。
2.1.3 把上层水相转移到新管中,用异丙醇沉淀水相中的RNA。每使用1 mL Trizol加入0.5 mL异丙醇,颠倒数次混匀,放置-20 ℃冰箱30 min。如果提取microRNA等小RNA,则置于-80 ℃沉淀过夜。
2.1.4 4 ℃ 12 000 rpm离心30 min,在管底可见RNA沉淀。
2.1.5 弃上清,用75%乙醇(DEPC水配制)洗涤RNA沉淀。每使用1 mL Trizo1至少加1 mL 75%乙醇。4 ℃, 7500 rpm离心5 min。
2.1.6 室温放置干燥RMA沉淀,大约晾3-5 min。加入适量无RNase的水溶解RNA后检测RNA浓度和纯度。
2.2 cDNA合成
反转录体系
试剂 | 使用量 (μL) |
RNA | ≤ 1 μg |
Enzyme Mix | 1 |
5×All-in-one qRT SuperMix | 4 |
RNase Free dH2O | Up to 20 |
上述反应体系在PCR仪50 ℃反应15 min进行逆转录,然后在85 ℃反应5 s使RT酶失活;得到的cDNA产物可保存于-80 ℃备用。
2.3 Real Time PCR
2.3.1 引物设计在NCBI上进行,需在实验前完成合成。
2.3.2 定量PCR反应体系
试剂 | 使用量 (μL) |
2×ChamQ Universal SYBR Qpcr Master | 10 |
Primer F(10uM) | 0.5 |
Primer R(10uM) | 0.5 |
Template DNA/cDNA | 1 |
ddH2O | To 20 |
2.3.3 通过上述体系混合配成MIX,加入qPCR管中混匀至管中无气泡。
2.3.4 两步法进行real time PCR,反应程序如下:
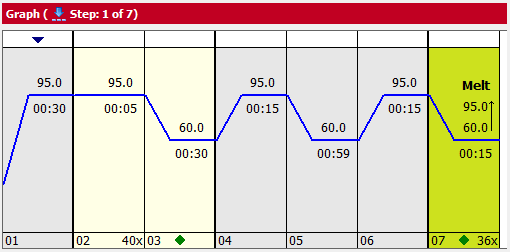
2.3.5 待反应结束后保存文件数据,数据分析使用双delta法(计算方式如下),结果表达形式为mean ± SD;使用单因素方差分析法比较两组数据间差异,P< 0.05为差异显著;∆Ct=目的基因Ct值-内参基因Ct值;∆∆Ct=各样品∆Ct-对照组∆Ct平均值;2-∆∆Ct反应各样品相对于照组目的基因的相对表达水平
3. 结果示例
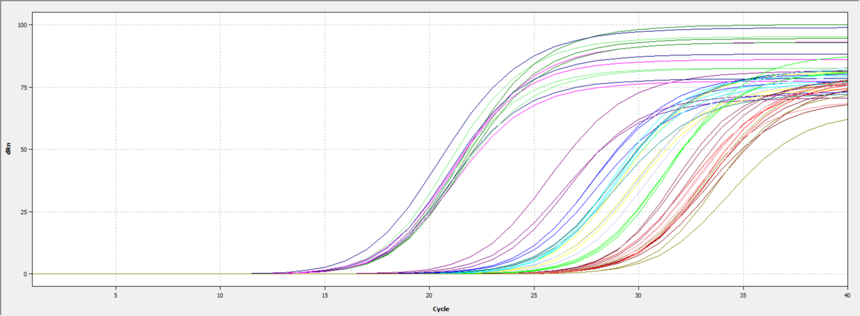
如上图所示为qPCR的扩增曲线,曲线越靠后,Ct值越大。
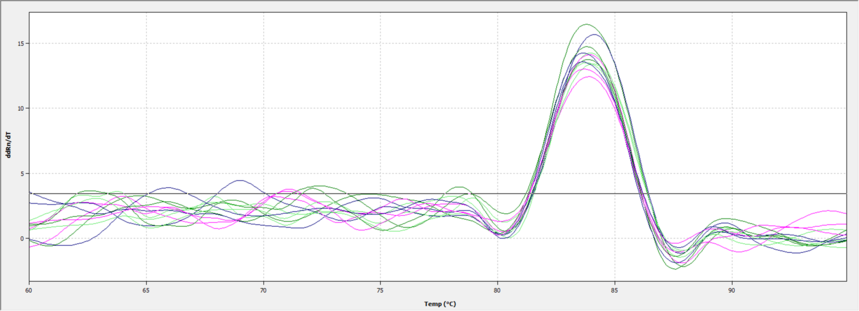
如上图所示为qPCR的溶解曲线,曲线单峰说明产物特异性高。
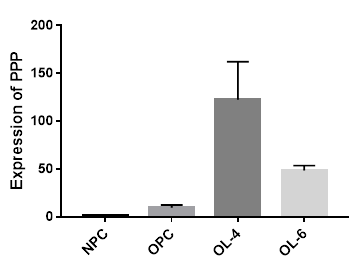
如上图所示为qPCR结果图,数值越高表明基因相对表达量越高。
参考文献
[1] Rio DC, Ares M Jr, Hannon GJ, Nilsen TW. Purification of RNA using TRIzol (TRI reagent). Cold Spring Harb Protoc. 2010 Jun;2010(6):pdb.prot5439.
[2] Rio DC, Ares M Jr, Hannon GJ, Nilsen TW. Nondenaturing agarose gel electrophoresis of RNA. Cold Spring Harb Protoc. 2010 Jun;2010(6):pdb.prot5445.
[3] Green MR, Sambrook J. Quantification of RNA by Real-Time Reverse Transcription-Polymerase Chain Reaction (RT-PCR). Cold Spring Harb Protoc. 2018 Oct 1;2018(10).
[4] Arya M, Shergill IS, Williamson M, Gommersall L, Arya N, Patel HR. Basic principles of real-time quantitative PCR. Expert Rev Mol Diagn. 2005 Mar;5(2):209-19.
[5] Thornton B, Basu C. Real-time PCR (qPCR) primer design using free online software. Biochem Mol Biol Educ. 2011 Mar-Apr;39(2):145-54.
[6] Guénin S, Mauriat M, Pelloux J, Van Wuytswinkel O, Bellini C, Gutierrez L. Normalization of qRT-PCR data: the necessity of adopting a systematic, experimental conditions-specific, validation of references. J Exp Bot. 2009;60(2):487-93.