肿瘤糖代谢的特征
根据文献【1】,肿瘤细胞独特的糖代谢特征是高糖吸收、有氧糖酵解(Warburg效应)与高乳酸生成,具体表现为肿瘤细胞参与氧化代谢的相关蛋白表达下调、葡萄糖转运蛋白及单羧酸转运蛋白表达上调。其实,这里就一个特点——有氧糖酵解,因为,高糖吸收和高乳酸生成是有氧糖酵解的上下游过程,没必要分成3个点!重点记住:肿瘤细胞的呼吸是即使有氧,也选择糖酵解!
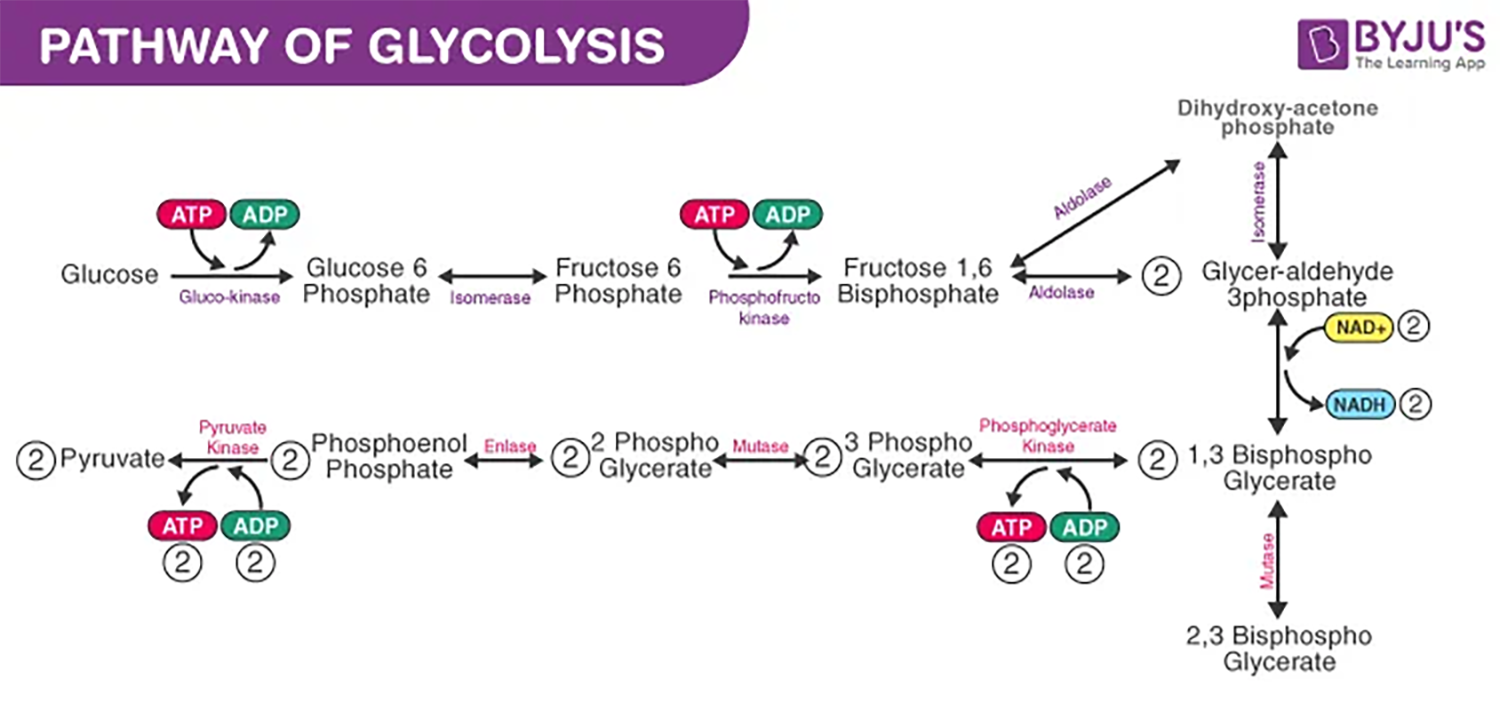
糖酵解途径
为什么有氧还要选择糖酵解?
DeepSeek是个好东西!让它来回答一下:

本来想着它把参考文献对应起来,方便我去核对文献是否正确,但是它很繁忙...每次都是多聊几句,它就忙不过来了。哎~幻方量化,你们赶紧增加算力啊!
回归正题,我们主要以第一个点(能量需求和代谢产物分流)为例,来检查一下DeepSeek的写作有没有问题。
上图文献1主要讲的是肿瘤细胞对有氧糖酵解的代谢物的吸收和整合,糖酵解速率增加没找到相关描述;

文献2讲了肿瘤微环境选择了糖酵解上调的肿瘤细胞,而有氧糖酵解可提供生物合成通路的底物。
文献3不存在,根据期刊名、卷、期、页信息找到的是一篇类风湿性关节炎的文章【2】,看来乱编文献的毛病仍然存在。
文献4讲的是谷氨酰胺代谢以及谷氨酰胺作为能量底物使细胞成瘾的一个基础研究。跟我们的问题相关性很弱!
文献5题目是Hallmarks of Cancer: The Next Generation,主要内容虽然不是糖酵解,但里面提到了肿瘤细胞通过增加葡萄糖吸收速率,解决了糖酵解产ATP不足的问题(如果是这样的话,糖酵解速率的提高可能不是原因,而是结果了;不过,也可能是与高效的葡萄糖吸收互为因果)。
文献6是2016年的一篇文章,相对新一点,关于DeepSeek提到的PI3K/Akt、c-MYC通路都有提及。根据以上这些内容,我们可以看出,AI生成的文章依然可能会编文献,依然会引用错误的文献,指令如果不是很清晰,它的话可能与参考文献难以一一对应。所以,直接让DeepSeek写综述,我们自己完全不带脑子,肯定是不行的。但DeepSeek确实可以写一个非常好的框架了,里面的具体内容需要我们一点点去找文献匹配起来。
综合以上全部内容,我们在回到问题——为什么有氧还要选择糖酵解?我的观点是肿瘤细胞重点是利用糖酵解的产物的增加(可能也包括有氧呼吸产物的减少)带来促癌作用;同时,因呼吸方式改变可能引起的ATP产量不足是可以通过增加葡萄糖的摄入来解决的。
糖酵解与哪些通路有关?
糖酵解的上游:主要受PI3K/Akt、AMPK、p53等通路调控或影响。

增殖细胞中活跃的代谢通路的上游:原癌基因和肿瘤抑制基因信号【3】。
糖酵解的下游(产物影响哪些通路):Pentose phosphate pathway(戊糖磷酸途径)、Hexosamine biosynthesis(己醣胺生物合成)、Glycerol-3 phosphate biosynthesis(甘油-3磷酸生物合成)、Serine/glycine biosynthesis One-carbon cycle(丝氨酸/甘氨酸生物合成 一碳循环),另外,据说排到细胞外的乳酸可以促进免疫抑制性肿瘤微环境的形成。

糖酵解产物主要用于生物合成和NADH合成【4】。
有氧糖酵解和无氧糖酵解的区别?
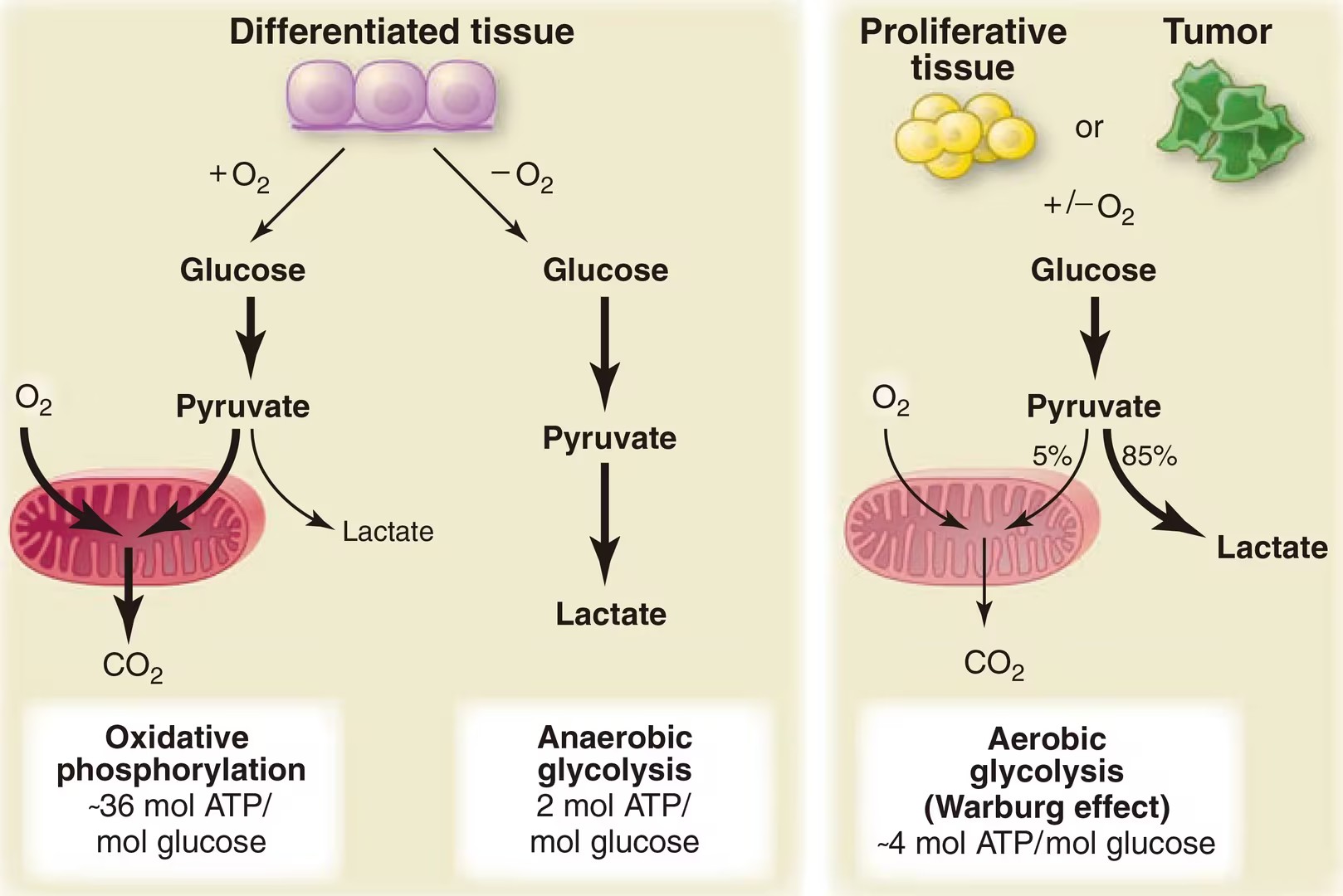
氧化磷酸化 vs 无氧糖酵解 vs 有氧糖酵解【3】
根据上面这个图,有氧糖酵解与无氧糖酵解的差别就是肿瘤细胞依然有少量的有氧呼吸。肿瘤细胞采用了牺牲氧化磷酸化的方式进行呼吸作用,但不是完全放弃了氧化磷酸化。如果在肿瘤细胞中完全阻断氧化磷酸化,或者大幅度调整丙酮酸进入有氧呼吸和无氧呼吸的比例,是否可以有效的抑制肿瘤细胞呢?这个问题就留给大家去从文献中查找吧!
参考文献
1. https://pmc.ncbi.nlm.nih.gov/articles/PMC10319507/
2. https://www.sciencedirect.com/science/article/abs/pii/S0006295201007079
3. https://www.science.org/doi/abs/10.1126/science.1160809
4. https://www.sciencedirect.com/science/article/pii/S155041311500621X#fig2